티스토리 뷰
728x90
반응형
문제
주어진 격자 데이터의 전후를 비교하여 주입된 백신이 CPCU-1202 백신인지 판단하는 문제이다.
CPCU-1202 백신을 놓으면, 백신이 주입된 조직에서 항체를 생성하고 같은 영역의 조직에 항체가 퍼진다.
항체는 조직을 변형시켜 동일한 색상이거나 다른 색상이 될 수 있다.
풀이
- 전체 조직의 영역들 중 색상이 바뀐 영역이 1개이하여야 한다.
- 따라서 조직을 탐색하면서 동일한 영역이 변경된 것인지를 판단하고, 색상이 변경된 영역의 개수를 세어야 한다.
- BFS나 DFS를 통해 동일한 색상으로 이루어진 인접 영역을 탐색하며 해당 영역이 일관된 색상으로 변경되었는지 확인한다.

- 탐색 중 영역이 다르다면 CPCU-1202 백신이 아님으로 NO를 출력하고,
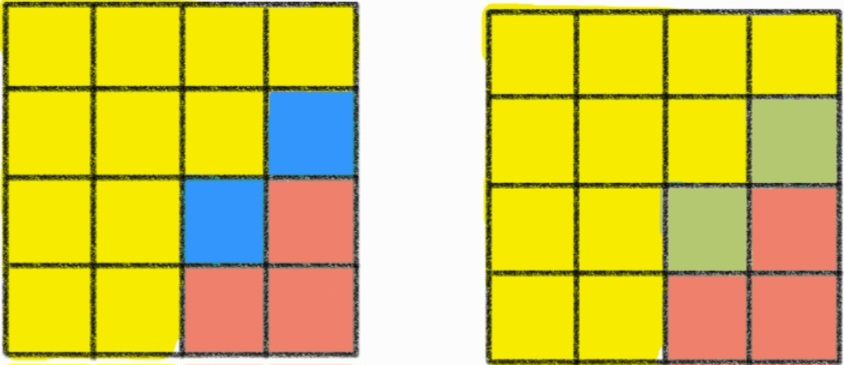
- 색상이 변경된 영역의 개수가 2개이상이어도 NO를 출력하며,

- 그 외의 경우는 CPCU-1202 백신임으로 YES를 출력한다.
Python 코드
import sys
input = sys.stdin.readline
n,m = map(int,input().split())
arr1 = [list(map(int,input().split())) for _ in range(n)]
arr2 = [list(map(int,input().split())) for _ in range(n)]
visited = [[0]*m for _ in range(n)]
def in_range(x,y):
return 0<=x<n and 0<=y<m
def check_area(i,j):
origin_color = arr1[i][j] # 기존 조직 색상
changed_color = arr2[i][j] # 변경된 조직 색상
stack = [(i,j)]
visited[i][j] = 1
while stack:
x, y = stack.pop()
for dx, dy in [(-1, 0), (1, 0), (0, -1), (0, 1)]:
nx, ny = x+dx, y+dy
if not in_range(nx,ny):
continue
if visited[nx][ny]:
continue
if arr1[nx][ny] == origin_color:
if arr2[nx][ny] == changed_color:
visited[nx][ny] = 1
stack.append((nx,ny))
else:
# 같은 값으로 업데이트 되어야만 CPCU-1202 백신
print("NO")
exit()
return origin_color == changed_color
cnt = 0
for i in range(n):
for j in range(m):
if not visited[i][j]:
if not check_area(i,j):
cnt += 1 # 변경된 영역
# 단 1개의 영역만 변해야함
if cnt <= 1:
print("YES")
else:
print("NO")
문제출처
728x90
반응형
'Study > Coding Test' 카테고리의 다른 글
| [백준 BOJ / Python] 1103번 게임 (0) | 2024.11.19 |
|---|---|
| [백준 BOJ / Python] 1113번 수영장 만들기 (0) | 2024.11.17 |
| [백준 BOJ / Python] 1743번 음식물 피하기 (1) | 2024.11.12 |
| [백준 BOJ / Python] 10942번 팰린드롬 (0) | 2024.10.12 |
| [프로그래머스 / Python & Java] PCCP 기출문제 1번 / 동영상 재생기 (1) | 2024.09.20 |
댓글
